Downloads
Download


This work is licensed under a Creative Commons Attribution 4.0 International License.
基于药效团拼接的贯叶连翘中橙酮类衍生物的设计、合成和抗3C-like protease活性研究
卫之雯,徐连华,彭语欣,兰嫒嫒,龙飞杨,张松林,李乔莉,王华斌*
贵州中医药大学药学院,贵州贵阳
摘要:目的:设计合成贯叶连翘中橙酮类结构新颖的衍生物并进行体外3C-like protease活性研究。方法:基于药效团拼接原理,选定贯叶连翘中橙酮类成分3,5,4'-三羟基橙酮类化合物作为母核结构,引入抗炎和抗病毒活性片段扑热息痛、霉酚酸和水杨酸、5-吲哚甲醛和布洛芬和含磷的药物以4或5碳原子链为连接链进行拼接,设计合成新型橙酮衍生物,并进行3C-like protease抑制活性和构效关系研究。结果:共设计、合成得到11个目标化合物,其中化合物9a对体外抗SARS-CoV-2 3CLpro活性作用最好。可以有效地抑制冠状病毒3C-like protease活性、抑制病毒的复制,具有潜在抗病毒作用。结论:研究结果表明化合物9a作为筛选出的目标药物,对抗3C-like protease活性极具潜力,可为贯叶连翘抗冠状病毒的进一步研究及药物开发提供科学依据。
冠状病毒(Coronavirus)是自然界常见的一类RNA病毒,分布广泛,且易变异,长期以来,冠状病毒已被认为是导致哺乳动物与鸟类呼吸道和胃肠道病变的重要病原体,对人体具有感染作用,会导致感冒、肺炎等疾病[1],并在全球范围内引起严重人类疾病传播,给人类健康带来了极大的威胁,同时也严重影响了世界经济发展。因此,抗冠状病毒药物的研发具有十分重要的意义。然而,现有的抗病毒药物仍不足以克服多种冠状病毒株带来的挑战,开发多靶点药物的方法在探索具有相似功能或结构的药物靶点方面受到限制。当前,越来越多的研究也表明药效团组合策略有利于克服以上这些缺点,由药效基团组合设计的单个药物分子可以作用于双重/多分子靶标[2]。例如,基于APN蛋白结构设计合成了多种盐霉素新型衍生物,揭示了其中的衍生物M1可以通过双靶向APN和3CL蛋白,有效抑制奥密克戎假病毒、猪流行性腹泻病毒等多种冠状病毒[3]。
过去的几十年中,越来越多的研究者通过对天然产物进行有效的活性修饰来提高其原有的生物活性。橙酮在自然界中的分布比较有限,大多存在于玄参科、菊科、苦苣苔科以及单子叶植物中的莎草科,在海洋中也偶尔能见到橙酮的身影。现代研究表明,它具有抗细胞毒性和肿瘤[4-6]、抗炎和抑菌[7]、乙酰胆碱酯酶抑制剂[8]、白细胞介素-5抑制剂[9]、抑制NO和PGE2产生[10]、SIRT1抑制剂[11]、ABCG2抑制剂[12,13]、17β-HSD1抑制剂[14]、抗氧化[15]等。如Hamdy等[16]发现类黄酮类化合物水飞蓟宾可通过多靶向活性显著干扰SARS-CoV-2的侵入和复制。橙酮类化合物作为具有较好的抗病毒、抗炎和抗菌的药效团,具有潜在的抗SARS-CoV-2病毒活性,最关键表现就是其可以抑制3CLpro的活性,3CLpro在介导SARS-CoV-2复制和转录中具有十分重要的作用,因此为了能够有效的抑制病毒的复制,可以尝试抑制3CLpro的活性来达到抗病毒的目的[17-18]。
另外,3CL蛋白酶(3CLpro)在冠状病毒的复制过程中有极其关键的作用,并且在突变株中有高度的结构相似性和保守性。因此,3CLpro已经成为了冠状病毒靶向药物研发的重要的靶标之一(图1)[19-22]。而橙酮类成分3,5,4'-三羟基橙酮类化合物作为3CL蛋白酶(3CLpro)结合位点抑制剂中的药效团,可以通过与3CL蛋白酶活性位点特异性结合,阻断酶与底物的结合,从而抑制3CL蛋白酶的活性,干扰病毒多聚蛋白的切割加工,阻断病毒的复制转录过程,达到抗病毒的效果。黄酮类化合物作为具有较好的抗病毒、抗炎和抗菌的药效团,具有潜在的抗SARS-CoV-2病毒活性。另外,扑热息痛、霉酚酸和水杨酸等药物和含磷的药物也具有良好的抗病毒、抗炎和抗菌活性[23-25],扑热息痛、霉酚酸和水杨酸等药物和含磷的药物也许可作为开发新的抗冠状病毒药物中一类重要的药效活性部分。因此,将橙酮类化合物和扑热息痛、霉酚酸和水杨酸等药物以及含磷的药物进行拼接,以期获得活性优良的抗冠状病毒药物。
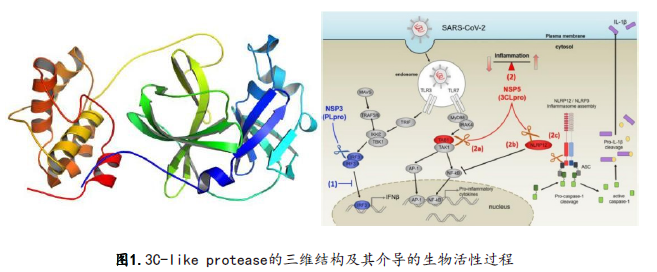
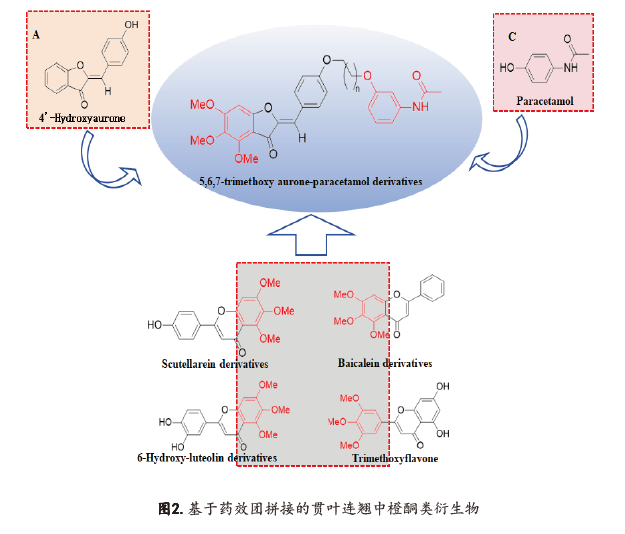
基于以上研究背景,本文将上述3,5,4'-三羟基橙酮类化合物与扑热息痛、霉酚酸和水杨酸、5-吲哚甲醛和布洛芬药物5种重要有效片段以及含磷的药物,利用有效的拼接组合方式共设计、合成得到了11个新型橙酮衍生物。具体是通过将三甲氧基苯基化学结构引入类橙酮的A环位置,进而与扑热息痛等通过不同长度的烷烃链进行连接(见图2)得到化合物8、5a~5e(图3)。另外通过将三甲氧基苯基化学结构引入类黄酮的A环位置,不同取代的二苯基氧磷和磷酸酯通过反应结合到一起,得到化合物9a~9e(图4)。最后对得到目标化合物8、5a~5e、9a~9e进行相关的体外抗3C-like protease活性研究。
1 材料
1.1 仪器
Waters Xevo G2-S QTOF质谱仪(美国Waters集团公司),JC-QX-10L超声波清洗器(青岛聚创环保集团有限公司),Waters ACQuity超高液相色谱仪(美国Waters集团公司),ACQUITY UPLCⓇ BEH C18 1.7 μm(2.1×100 mm)(美国Waters集团公司)、酶标仪(帝肯infinite Eplex)、细胞孵育箱。
1.2 试药
甲醇(AR,天津市富宇精细化工有限公司)、乙腈(GR,阿拉丁试剂有限公司)、甲酸(GR,阿拉丁试剂有限公司)。新型冠状病毒3CLpro蛋白酶抑制剂筛选试剂盒(碧云天)(https://www.beyotime.com/product/P0315M.htm)、DMSO(分析纯)、96孔细胞培养板(黑底黑盖)。
2 方法与结果
2.1 目标化合物合成路线
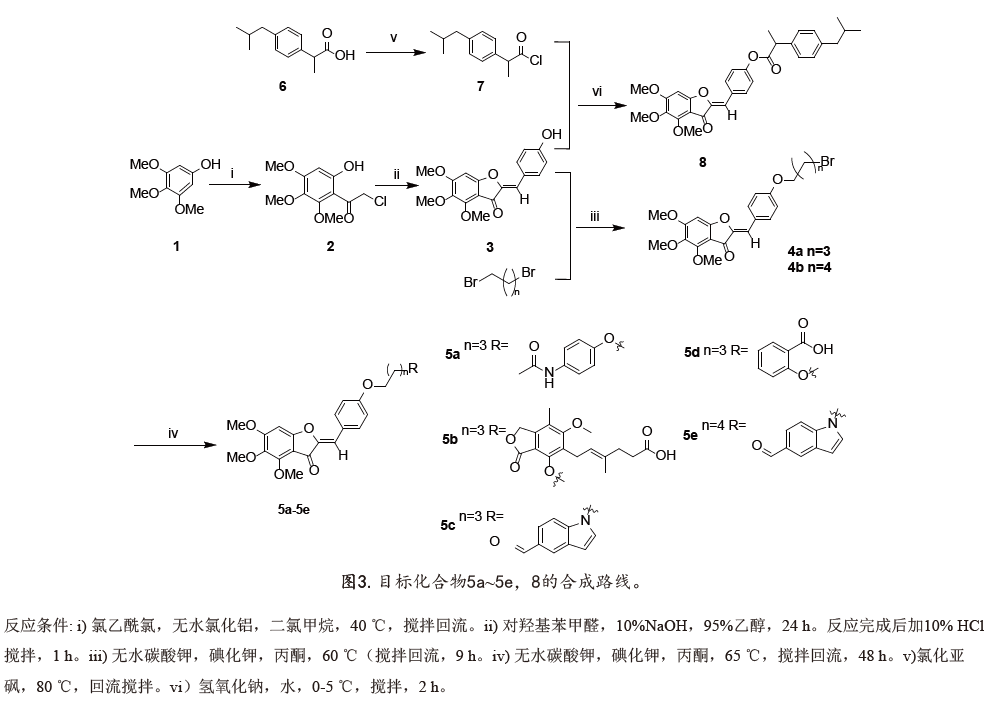
2.1.1 目标化合物5a~5e、8的合成
以3,4,5-三甲氧基苯酚为原料,经傅克反应,与对羟基苯甲醛环合,得到中间体化合物3。将化合物3与不同碳数的溴烃通过取代反应连接,然后和不同的药物片段结合,得到目标化合物5a~5e。将布洛芬酰氯化然后与化合物3结合得到目标化合物8(图3)。
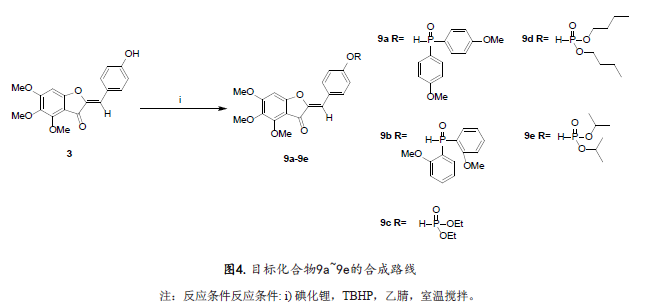
2.1.2 目标化合物9a~9e的合成
将化合物3与不同取代的二苯基氧磷和磷酸酯通过反应结合到一起, 得到目标化合物9a~9e(图4)。
2.2 目标化合物的合成
2.2.1 中间体4'-羟基-5,6,7-三甲氧基橙酮(3)的合成
称取3,4,5-三甲氧基苯酚(1)(3.68g,20mmol)溶解在40mL二氯甲烷中,加入无水氯化铝(2.67g,19.9mmol),缓慢滴加2mL氯乙酰氯,在40℃温度下搅拌回流反应约3h,TLC监测反应进程,待反应完毕,将反应液倒入V(浓盐酸):V(冰水)=(1:1)得溶液中,剧烈搅拌,用乙酸乙酯(50mL)萃取三次,无水硫酸钠除水后旋干,通过硅胶柱纯化,洗脱剂为石油醚:乙酸乙酯=40:1,即可得到片状黄白色沉淀6-羟基-2,3,4-三甲氧基-a-氯代苯乙酮(2)(2.30g),收率为44.2%。将中间体化合物2与对羟基苯甲醛(1.25g,10.24mmol)经氢氧化钠催化生成查尔酮,进而在室温条件下,在10%NaOH和95%乙醇的溶剂中闭合环化,得到粗产物中间体4'-羟基-5,6,7-三甲氧基橙酮(3)并在95%乙醇中重结晶,得到黄色粉末固体(1.9g,5.79mmol),收率为85.6%。
2.2.2 目标化合物5a~5e的合成
将中间体4a(0.10 g,0.22 mmol)和扑热息痛(0.04 g,0.26 mmol)溶解在5 mL丙酮中,边搅拌边向溶液中加入碳酸钾和碘化钾,在65 ℃下搅拌回流反应约48 h,经检测反应完成后,直接真空蒸发溶剂,利用硅胶快速色谱柱纯化,用洗脱剂石油醚:乙酸乙酯=1:1洗脱纯化,重结晶得到黄色粉末状目标化合物5a。化合物5b~5e以类似的方法得到。
2.2.3 目标化合物8的合成
将化合物7(0.11 g,0.5 mmol)在0-5 ℃下加入到10%氢氧化钠水溶液中(2 mL)中,在0-5 ℃下搅拌反应2 h,通过TLC监测反应,反应完成后,用乙酸乙酯(20 mL)萃取三次后,无水硫酸钠处理,然后真空浓缩,残余物通过硅胶快速色谱柱纯化,洗脱剂选用石油醚:乙酸乙酯=10:1,得目标化合物8。
2.2.4 目标化合物9a~9e的合成
将化合物3(0.082 g,0.25 mmol)溶解到2 mL乙腈中,再向该混合溶液中加入碘化锂(0.034 g,0.25 mmol)和TBHP(0.103 mL,0.75 mmol),最后再加入二苯基氧磷(0.051 g,0.25 mmol),在室温下反应1h,通过TLC监测反应,反应完成后,将反应液用乙酸乙酯萃(30 mL)取分层后,有机相用无水硫酸钠处理,然后浓缩,残余物通过硅胶快速色谱柱纯化,用洗脱剂V石油醚:V乙酸乙酯(5:1)洗脱纯化,得到黄色粉末状目标化合物9a。化合物9b~9e以类似的方法得到。
2.3 目标化合物的谱图数据
2.3.1 目标化合物5a
黄色粉末,收率76%;1H NMR (400 MHz, DMSO-d6) δ 9.76 (s, 1H), 7.92–7.89 (m, 2H), 7.48–7.44 (m, 2H), 7.07–7.04 (m, 2H), 6.97 (s, 1H), 6.88–6.83 (m, 2H), 6.75 (s, 1H), 4.10 (s, 5H), 3.99 (t, J = 5.8 Hz, 2H), 3.95 (s, 3H), 3.68 (s, 3H), 1.99 (s, 3H), 1.89–1.85 (m, 4H); 13C NMR (101 MHz, Chloroform-d) δ 180.94, 168.41, 163.94, 161.56, 160.26, 155.89, 151.70, 146.68, 136.67, 133.12, 131.14, 125.25, 122.03, 115.00, 114.85, 111.74, 107.41, 90.6·7, 67.76, 67.70, 62.51, 61.76,56.74, 26.06, 26.04, 24.47; HRMS (ESI) calcd for C30H31NO8 [M+H]+: 534.2122, found 534.2123.
2.3.2 目标化合物5b
黄色粉末,收率33%;1H NMR (400 MHz, DMSO-d6) δ 9.34 (s, 1H), 7.90–7.86 (m, 2H), 7.04–6.95 (m, 2H), 6.95 (s, 1H), 6.73 (s, 1H), 5.21 (s, 2H), 5.13 (td, J = 6.3, 5.8, 3.1 Hz, 1H), 4.10 (s, 3H), 4.05–3.97 (m, 4H), 3.95 (s, 3H), 3.68 (d, J = 4.9 Hz, 6H), 3.27 (d, J = 6.9 Hz, 2H), 2.35 (t, J = 7.7 Hz, 2H), 2.18 (t, J = 7.4 Hz, 2H), 2.05 (s, 3H), 1.75–1.65 (m, 7H); 13C NMR (101 MHz, Chloroform-d) δ 180.84, 173.55, 173.05, 163.88, 163.72, 161.49, 160.23, 153.68, 151.65, 146.62, 144.11, 136.61, 134.24, 133.05, 125.16, 122.76, 122.18, 116.84, 114.92, 111.63, 107.35, 106.41, 90.62, 70.16, 67.82, 64.23, 62.50, 61.74, 61.09, 56.72, 34.73, 33.16, 28.44, 22.67, 16.22, 11.65; HRMS (ESI) calcd for C39H42O12 [M+H]+: 703.2749, found 703.2745.
2.3.3 目标化合物5c
黄色粉末,收率83%;1H NMR (400 MHz, Chloroform-d) δ 10.01 (s, 1H), 8.15 (d, J = 1.6 Hz, 1H), 7.81–7.77 (m, 3H), 7.43 (d, J = 8.7 Hz, 1H), 7.22 (d, J = 3.2 Hz, 1H), 6.91–6.87 (m, 2H), 6.72 (s, 1H), 6.66 (dd, J = 3.2, 0.8 Hz, 1H), 6.53 (s, 1H), 4.27–4.24 (m, 5H), 4.00–3.95 (m, 5H), 3.81 (s, 3H), 2.11–2.04 (m, 2H), 1.84–1.77 (m, 2H); 13C NMR (101 MHz, Chloroform-d) δ 192.56, 180.83, 163.88, 161.54, 159.91, 151.66, 146.71, 139.35, 136.66, 133.06, 129.76, 129.41, 128.43, 126.76, 125.44, 121.81, 114.88, 111.43, 110.01, 107.32, 103.62, 90.63, 67.37, 62.48, 61.71, 56.70, 46.45, 27.19, 26.59; HRMS (ESI) calcd for C32H31NO7 [M+H]+: 542.2173, found 542.2175.
2.3.4 目标化合物5d
黄色粉末,收率84%;1H NMR (400 MHz, Chloroform-d) δ 10.82 (s, 1H), 7.83–7.79 (m, 3H), 7.47–7.41 (m, 1H), 6.99–6.93 (m, 3H), 6.88–6.84 (m, 1H), 6.74 (s, 1H), 6.54 (s, 1H), 4.45–4.43 (m, 2H), 4.25 (s, 3H), 4.11–4.05 (m, 2H), 3.97 (s, 3H), 3.82 (s, 3H), 2.05–1.94 (m, 4H); 13C NMR (101 MHz, Chloroform-d) δ 180.87, 170.28, 163.91, 161.79, 161.53, 160.06, 151.71, 146.71, 136.68, 135.84, 133.10, 129.92, 125.40, 119.26, 117.72, 114.96, 112.53, 111.56, 107.39, 90.64, 67.39, 65.05,62.51, 61.74, 56.71, 25.93, 25.50; HRMS (ESI) calcd for C29H28O9 [M+H]+: 521.1806, found 521.1809.
2.3.5 目标化合物5e
黄色粉末,收率80%;1H NMR (400 MHz, DMSO-d6) δ 9.97 (s, 1H), 8.18 (d, J = 1.2 Hz, 1H), 7.90–7.86 (m, 2H), 7.67 (d, J = 1.2 Hz, 2H), 7.57 (d, J = 3.2 Hz, 1H), 7.02–6.97 (m, 3H), 6.75 (s, 1H), 6.68 (d, J = 3.1 Hz, 1H), 4.27 (t, J = 7.0 Hz, 2H), 4.10 (s, 3H), 4.03–3.97 (m, 2H), 3.95 (s, 3H), 3.68 (s, 3H), 1.89–1.72 (m, 4H), 1.44–1.38 (m, 2H); 13C NMR (101 MHz, Chloroform-d) δ 192.54, 180.80, 163.85, 161.50, 160.07, 151.62, 146.64, 139.31, 136.62, 133.02, 129.82, 129.32, 128.38, 126.67, 125.25, 121.75, 114.88, 111.49, 109.97, 107.30, 103.46, 90.62, 67.61, 62.46, 61.69, 56.68, 46.61, 30.06, 28.78, 23.59; HRMS (ESI) calcd for C31H29NO7 [M+H]+: 528.2017, found 534.2019.
2.3.6 目标化合物8
黄色粉末,收率77%;1H NMR (400 MHz, Chloroform-d) δ 7.85–7.81 (m, 2H), 7.34–7.29 (m, 2H), 7.16–7.14 (m, 2H), 7.10–7.04 (m, 2H), 6.72 (s, 1H), 6.52 (s, 1H), 4.24 (s, 3H), 3.99–3.93 (m, 4H), 3.81 (s, 3H), 2.47 (d, J = 7.2 Hz, 2H), 1.94–1.81 (m, 1H), 1.61 (d, J = 7.1 Hz, 3H), 0.91 (d, J = 6.6 Hz, 6H); 13C NMR (101 MHz, Chloroform-d) δ 180.85, 173.06, 164.10, 161.81, 151.65, 147.63, 141.02, 137.12, 136.74, 132.31, 130.22, 129.66, 129.45, 127.30, 121.99, 110.34, 107.04, 90.65, 62.46, 61.69, 56.73, 45.37, 45.12, 30.27, 30.24, 22.48, 18.58; HRMS (ESI) calcd for C31H32O7 [M+H]+: 517.2221, found 517.2223.
2.3.7 目标化合物9a
黄色粉末,收率73%;1H NMR (400 MHz, Chloroform-d) δ 7.82–7.69 (m, 6H), 7.26–7.22 (m, 2H), 6.93 (dq, J = 9.4, 2.7 Hz, 4H), 6.63 (s, 1H), 6.49 (s, 1H), 4.21 (s, 3H), 3.93 (s, 3H), 3.79 (d, J = 5.5 Hz, 9H); 13C NMR (101 MHz, Chloroform-d) δ 180.74, 163.98, 162.93 (d, J = 3.4 Hz), 161.72, 151.98 (d, J = 8.1 Hz), 151.68, 147.34, 136.66, 133.70 (d, J = 11.8 Hz), 132.70, 128.89, 122.90, 121.23 (d, J = 4.9 Hz), 114.25 (d, J = 14.6 Hz), 110.36, 106.99, 90.62, 62.40, 61.65, 56.70, 55.40; HRMS (ESI) calcd for C32H29O9P [M+H]+: 589.1622, found 589.1624.
2.3.8 目标化合物9b
黄色粉末,收率66%;1H NMR (400 MHz, Chloroform-d) δ 8.00 (ddd, J = 14.2, 7.6, 1.8 Hz, 2H), 7.73–7.70 (m, 2H), 7.52–7.46 (m, 2H), 7.28–7.24 (m, 2H), 7.10–7.02 (m, 2H), 6.88–6.83 (m, 2H), 6.66 (s, 1H), 6.51 (s, 1H), 4.23 (s, 3H), 3.96 (s, 3H), 3.80 (s, 3H), 3.64 (s, 6H); 13C NMR (101 MHz, Chloroform-d) δ 180.89, 164.06, 161.72, 161.17 (d, J = 4.4 Hz), 152.44 (d, J = 8.0 Hz), 151.78, 147.28, 136.73, 134.89 (d, J = 6.7 Hz), 134.36, 132.57, 128.66, 121.37 (d, J = 5.1 Hz), 120.58 (d, J = 13.0 Hz), 120.26, 118.85, 111.35 (d, J = 8.1 Hz), 110.80, 107.16, 90.67, 62.50, 61.74, 56.76, 55.76; HRMS (ESI) calcd for C32H29O9P [M+H]+: 589.1622, found 589.1624.
2.3.9 目标化合物9c
黄色粉末,收率70%;1H NMR (400 MHz, DMSO-d6) δ 8.00–7.96 (m, 2H), 7.34–7.30 (m, 2H), 6.96 (s, 1H), 6.78 (s, 1H), 4.22–4.14 (m, 4H), 4.10 (s, 3H), 3.95 (s, 3H), 3.69 (s, 3H), 1.28 (td, J = 7.1, 1.0 Hz, 6H); 13C NMR (101 MHz, Chloroform-d) δ 180.68, 163.97, 161.71, 151.64, 151.34 (d, J = 6.7 Hz), 147.43, 136.60, 132.66, 129.39, 120.35 (d, J = 5.2 Hz), 110.00, 106.88, 90.57, 64.78 (d, J = 6.1 Hz), 62.35, 61.59, 56.66, 16.09 (d, J = 6.7 Hz); HRMS (ESI) calcd for C22H25O9P [M+H]+: 465.1309, found 465.1311.
2.3.10 目标化合物9d
黄色粉末,收率75%;1H NMR (400 MHz, ) δ 7.85–7.81 (m, 2H), 7.29–7.25 (m, 2H), 6.70 (s, 1H), 6.53 (s, 1H), 4.23 (s, 3H), 4.19–4.11 (m, 4H), 3.96 (s, 3H), 3.80 (s, 3H), 1.71–1.63 (m, 4H), 1.44–1.35 (m, 4H), 0.91 (t, J = 7.4 Hz, 6H); 13C NMR (101 MHz, Chloroform-d) δ 180.92, 164.15, 161.86, 151.87, 147.60, 136.81, 132.80, 131.06, 129.54, 128.97, 120.50 (d, J = 5.1 Hz), 110.26, 107.12, 90.69, 68.62 (d, J = 6.5 Hz), 62.53, 61.77, 56.80, 32.31 (d, J = 6.6 Hz), 30.68, 29.82, 18.73, 13.66; HRMS (ESI) calcd for C26H33O9P [M+H]+: 521.1935, found 521.1937.
2.3.11 目标化合物9e
黄色粉末,收率71%;1H NMR (400 MHz, Chloroform-d) δ 7.85–7.82 (m, 2H), 7.30–7.26 (m, 2H), 6.72 (s, 1H), 6.54 (s, 1H), 4.77 (dq, J = 12.7, 6.3 Hz, 2H), 4.24 (s, 3H), 3.97 (s, 3H), 3.81 (s, 3H), 1.37 (d, J = 6.2 Hz, 6H), 1.32 (d, J = 6.2 Hz, 6H); 13C NMR (101 MHz, Chloroform-d) δ 180.79, 164.02, 161.72, 151.73 (d, J = 3.4 Hz), 147.43, 136.69, 132.63, 129.21, 120.46 (d, J = 5.6 Hz), 110.25, 107.02, 90.58,73.80(d, J = 6.4 Hz), 62.41, 61.65, 56.68, 23.62 (dd, J = 10.2, 5.1 Hz); HRMS (ESI) calcd for C24H29O9P [M+H]+: 493.1622, found 49.1624.
2.4 贯叶连翘中橙酮类衍生物抗3C-like protease活性
2.4.1 抗病毒活性实验
2.4.1.1 样品的准备
1)取适量待测定的样品,用DMSO等适当的溶剂配制成所需浓度和浓度梯度的溶液。
2)根据样品的数量配制检测试剂(Assay Reagent),每个样品需1 μL 2019-nCov 3CLpro和92 μL检测缓冲液(Assay Buffer)。
2.4.1.2 样品测定
进行样品测定之前,先将Assay Reagent和3CLpro配置成所需的Assay Buffer,每份样品需要93 μL Assay Buffer,其中包含92 μL Assay Reagent和1 μL 3CLpro,然后设置不同的组,如下:
1)空白对照组每孔需要加入93 μL Assay Buffer和5 μL DMSO;
2)100%酶活性对照组每孔需要加入93 μL Assay Reagent和5 μL DMSO;
3)阳性对照组每孔需要加入93 μL Assay Reagent和5 μL奈玛特韦DMSO溶液;
4)目标化合物组每孔需要加入93 μL Assay Reagent和5 μL目标化合物溶液(5a~5e,9a~9e,8)。
每一个浓度均设置2个复孔。将这些样品加入到全黑的96孔板中,然后各孔快速加入Substrate 2 μL,混匀,盖上盖子后于37 ℃的恒温环境中孵育5 min,孵育结束后,立马使用多功能酶标仪进行荧光值测定(激发波长340 nm,发射波长490 nm)。
2.4.2 抑制率计算
1)计算每个空白对照孔、100%酶活性对照孔和样品孔的平均荧光值(RFU),可分别记录为RFU空白对照、RFU100%酶活性对照和RFU样品。
2)计算每个样品的抑制百分率。
计算公式:抑制率(%)=(RFU100%酶活性对照-RFU样品)/(RFU100%酶活性对照-RFU空白对照)。
3)利用GraphPad Prism 8软件计算化合物对蛋白酶的抑制率,结果见表1。
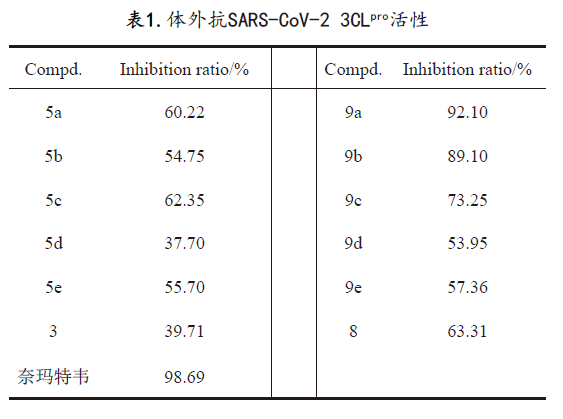
aInhibitory activities at 500 μmol·L-1。
2.5 结果
根据蛋白酶活性测试结果可知,在500 μmol·L-1浓度下,中间体化合物3在对SARS CoV-2 3CLpro抑制率较低,仅为39.71%。因此,首先对中间体化合物3进行结构修饰,从而得到了一系列衍生物5a~5e,8和9a~9e,实验结果显示修饰后的化合物相较于中间体化合物3,其活性确实有所提升,显示结果数据较好,其中活性最好的是9a,抑制率为92.10%。
3 结论
本研究基于药效团拼合原理,以具有抑制SARS-CoV-2病毒复制活性的橙酮类化合物作为母核结构,通过引入对其维持合适的药效团如扑热息痛、霉酚酸和水杨酸、5-吲哚甲醛和布洛芬药物5种重要有效片段以及含磷的药物,利用有效的拼接组合方式共设计、合成得到了11个新型橙酮衍生物,并通过核磁共振、质谱等波谱手段进行了结构表征。通过体外酶活性实验测定这些化合物对SARS-CoV-2 3CL蛋白酶的抑制活性,结果显示含磷及水杨酸片段的橙酮类衍生物表现出显著的抑制效果,其中化合物9a对3CLpro的抑制活性最为突出。该结果表明此类结构可通过抑制SARS-CoV-2 3CLpro活性发挥潜在抗冠状病毒作用,值得进一步深入科学研究。
因此,贯叶连翘作为我国传统的中药材,具有良好的前景,我们通过研究发现,贯叶连翘提取物中的橙酮类化合物的衍生物具有一定抗SARS-CoV-2活性,但具体构效关系的研究还不够深入,在后续的研究中,将继续开发新的橙酮类衍生物,测试SARS-CoV-2相关靶点,深入研究构效关系,以期获得具有良好的抗SARS-CoV-2单体化合物,为贯叶连翘的资源开发和抗冠状病毒新药的研发奠定基础。
参考文献
[2] Raghavendra NM, Pingili D, Kadasi S, et al. Dual or multi-targeting inhibitors:The next generation anticancer agents[J]. Eur J Med Chem. 2018, 143: 1277-1300.
[3] Zheng YL, Feng J, Song YB, et al. Design and synthesis of APN and 3CLpro dual-target inhibitors based on STSBPT with anticoronavirus activity[J]. Medical Virology, 2024, 96(3): e29512.
[4] Huang W, Liu MZ, Li Y, et al. Design, syntheses, and antitumor activity of novel chromone and aurone derivatives[J]. Bioorg Med Chem. 2007, 15: 5191.
[5] Lawrence NJ, Rennison D, McGown AT, et al. The total synthesis of an aurone isolated from Uvaria hamiltonii: aurones and flavones as anticancer agents[J]. Bioorg Med Chem Lett. 2003, 13: 3759.
[6] Lewin G, Aubert G, Thoret S, et al. Influence of the skeleton on the cytotoxicity of flavonoids[J]. Bioorg Med Chem. 2012, 20:1231.
[7] Bandgar BP, Patil SA, Korbad BL, et al. Synthesis and biological evaluation of a novel series of 2,2-bisaminomethylated aurone analogues as anti-inflammatory and antimicrobial agents[J]. Eur J Med Chem. 2010 Jul;45(7):3223-3227.
[8] Sheng R, Xu Y, Hu CQ, et al. Design, synthesis and AChE inhibitory activity of indanone and aurone derivatives[J]. Eur J Med Chem. 2009 Jan;44(1):7-17.
[9] Thanigaimalai P, Yang HM, Sharma VK, et al. The scope of thallium nitrate oxidative cyclization of chalcones; synthesis and evaluation of isoflavone and aurone analogs for their inhibitory activity against interleukin-5[J]. Bioorg Med Chem. 2010 Jun 15;18(12):4441-4445.
[10] Shin SY, Shin MC, Shin JS, et al. Synthesis of aurones and their inhibitory effects on nitric oxide and PGE2 productions in LPS-induced RAW 264.7 cells[J]. Bioorg Med Chem Lett. 2011 Aug 1;21(15):4520-4523.
[11] Manjulatha K, Srinivas S, Mulakayala N, et al. Ethylenediamine diacetate (EDDA) mediated synthesis of aurones under ultrasound: their evaluation as inhibitors of SIRT1[J]. Bioorg Med Chem Lett. 2012 Oct 1;22(19):6160-6165.
[12] Sim HM, Wu CP, Ambudkar SV, et al. In vitro and in vivo modulation of ABCG2 by functionalized aurones and structurally related analogs[J]. Biochem Pharmacol. 2011 Dec 1;82(11):1562-1571.
[13] Sim HM, Lee CY, Ee PL, et al. Dimethoxyaurones: Potent inhibitors of ABCG2 (breast cancer resistance protein)[J]. Eur J Pharm Sci. 2008 Nov 15;35(4):293-306.
[14] Starčević Š, Turk S, Brus B, et al. Discovery of highly potent, nonsteroidal 17β-hydroxysteroid dehydrogenase type 1 inhibitors by virtual high-throughput screening[J]. J Steroid Biochem Mol Biol. 2011 Nov;127(3-5):255-261.
[15] Detsi A, Majdalani M, Kontogiorgis CA, et al. Natural and synthetic 2'-hydroxy-chalcones and aurones: synthesis, characterization and evaluation of the antioxidant and soybean lipoxygenase inhibitory activity[J]. Bioorg Med Chem. 2009 Dec 1;17(23):8073-8085.
[16] Hamdy R, Mostafa A, Abo Shama NM, et al. Comparative evaluation of flavonoids reveals the superiority and promising inhibition activity of silibinin against SARS-CoV-2[J]. Phytother Res. 2022, 36(7): 2921-2939.
[17] Zhang LL, Lin DZ, Sun XYY, et al. Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors[J]. Science, 2020, 368(6489): 409-412.
[18] Xiong MY, Su HX, Zhao WF, et al. What coronavirus 3C‐like protease tells us: from structure, substrate selectivity, to inhibitor design[J]. Medicinal Research Reviews, 2021, 41(4): 1965-1998.
[19] 闵瑞, 刘洁, 代喆等. 新型冠状病毒肺炎发病机制及临床研究进展[J]. 中华医院感染学杂志, 2020, 30 (08): 1171-1176.
[20] Zhou P, Yang XL, Wang XG, et al. A pneumonia outbreak associated with a new coronavir us of probable bat origin[J]. Nature, 2020, 579: 270-273.
[21] Anand K, Ziebuhr J, Wadhwani P, et al. Coronavirus main proteinase (3CLpro) structure: Basis for design of antiSARS drugs[J]. Science, 2003, 300: 1763-1767.
[22] Yang HT, Yang MJ, Ding Y, et al. The crystal structures of severe acute respiratory syndrome virus main protease and its complex with an inhibitor[J]. Proc Natl Acad Sci. USA, 2003, 100: 13190-13195.
[23] Li J, Chen L, Li C, et al. Phosphorus dendron nanomicelles as a platform for combination anti-inflammatory and antioxidative therapy of acute lung injury[J]. Theranostics, 2022, 12: 3407-3419.
[24] Kato F, Matsuyama S, Kawase M, et al. Antiviral activities of mycophenolic acid and IMD-0354 against SARS-CoV-2[J]. Microbiol Immunol. 2020, 64: 635-639.
[25] Jamshidinia N, Saadatpour F, Arefian E, et al. Augmented antiviral activity of chlorhexidine gluconate on herpes simplex virus type 1, H1N1 influenza A virus, and adenovirus in combination with salicylic acid[J]. Arch Virol. 2023, 168: 302.